摘要
- 随着医学成像技术和计算机技术的不断发展和进步,医学图像分析已成为医学研究、临床疾病诊断和治疗中一个不可或缺的工具和技术手段。近几年来,深度学习(Deep learning,DL),特别是深度卷积神经网络(Convolutional neural networks,CNNs)已经迅速发展成为医学图像分析的研究热点,它能够从医学图像大数据中自动特区隐含的疾病诊断特征。本文首先简述医学图像分析的特点;其次,论述深度学习的基本原理,介绍主要用于医学图像分析的主要深度学习技术;然后分别论述深度学习在医学图像分类、检测、分割、配准、检索、图像生成和增强等各领域的国内外研究现状;最后,讨论归纳医学图像分析深度学习方法未来要面临的挑战和应对的策略。
- 关键词:医学图像分析;深度学习;卷积神经网络
1、介绍
- 在医学图像领域,医生或者研究人员在对某种特定的内部组织器官进行定量分析、实时监控和治疗规划时,为了做出正确的治疗决策,通常需要了解这种组织器官的一些详细信息。所以生物医学影像已成为疾病诊断和治疗中不可或缺的组成部分,且日益重要。
- 核磁共振成像 (Magnetic resonance image, MRI)、正电子发射断层扫 描 (Positron emission tomography, PET)、计算机 断层扫描 (Computer tomography, CT)、锥形束 CT、3D 超声成像等医学影像技术目前已广泛应用于临床检查、诊断、治疗与决策。如何充分利用人工智能深度学习方法分析处理这些超大规模的医学图像大数据,为临床医学中各种重大疾病的筛查、诊断、疗效评估提供科学方法,是当前医学图像分析领域急需解决的重大科学问题和前沿医学影像关键技术。
- 医学图像分析最初主要采用边缘检测、纹理特征、形态学滤波以及构建形状模型和模板匹配等方法。这类分析方法通常针对特定任务而设计,被称为手工定制式设计方法。而深度学习是以数据驱动方式分析任务,能自动地从特定问题的大规模数据集中学习相关模型特征和数据特性。与针对特定问题而显式地手工设计模型不同,深度学习方法可直接从数据样本中隐式地自动学习医学图像特征,其学习过程本质上是一个优化问题的求解过程。通过学习,模型从训练数据中选择正确的特征,使其在测试新数据时做出正确决策。因此,深度学习在医学图像 分析中起着至关重要的作用。
- 近年来, 深度学习不断取得重大进展, 主要得益于不断提高的计算能力和持续增长的可用数据量,以及深度学习模型及其算法的不断改进。其实质是通过构建多隐层的机器学习模型,利用海量的样本数据训练,学习更精准的特征,最终提高分类或预测的准确性[1]。 深度学习这种从数据中学习层次特征的特点,使得它非常适合发现高维数据中的复杂结构,目前深度学习已经应用到图像识别、语音识别、自然语言处理、天气预测、基因表达、内容推荐等领域和各种挑战赛中。
- 深度学习在计算机视觉领域的巨大成功, 激发了国内外许多学者将其应用于医疗图像分析。近年来,已有多位专家对深度学习在医学图像分析中的研究现状及问题进行了总结、评述和讨论。最近,Medical Image Analysis上发表的综述对深度学习在医学图像分类、检测和分割、配准 和检索等方面的研究进行了较全面的归纳总结[2] 。
2、医学图像分析的特点
- 医学图像分析已广泛应用于良恶性肿瘤、脑功能与精神障碍、心脑血管疾病等重大疾病的临床辅助筛查、诊断、分级、治疗决策与引导、疗效评估等方面。医学图像分类与识别、定位与检测、组织器官与病灶分割是当前医学图像分析深度学习方法研究主要应用领域。不同成像原理的医学图像分析和计算机视觉领域中的自然图像分析存在较大的差别。至今为止,国内外学者主要针对MRI、CT、X射线、超声、PET、病理光学显微镜等不同成像原理的医学图像分析任务开展了一系列的深度学习研究工作。因此,本节主要简述这几种医学图像的分析。
2.1 常见的医学图像
- MRI图像:核磁共振图像(MRI),该图像是人体组织器官和病灶中的氢原子核在外部强磁场作用下产生的磁共振信号大小的度量,并通过计算机对体外核磁共振信号探测器接收到的信息数据进行3D图像重建。它能够提供非常清晰的人体软组织解剖结构和病灶影像。
- CT图像:计算机断层扫描(CT)利用精确准直的X射线束对人体某部位一定厚度的断面进行照射扫描,并由与射线线束一起旋转的探测器接收透射穿过该断面的X射线,最后,计算机根据探测器接收到的X射线信号数据重建相应人体断面的3D图像。它具有亚毫米级的空间分辨率,能够提供清晰的人体骨性组织解剖结构和病灶影像,已广泛应用于多种临床疾病检查和辅助诊断。
- X射线图像:医学X射线图像是人体不同组织器官和病灶的电子密度度量影像。基于X射线的成像包括2D的计算机放射成像、数字化 X 射线摄影术、数字减影血管造影术和乳房X线摄影术,以及3D的螺旋计算机断层扫描术等,已广泛地应用于骨科、肺部、乳腺和心血管等临床疾病检测和辅助诊断,但2DX射线图像不能提供人体组织器官和病灶的三维立体信息。
- 超声成像:利用超声束扫描人体,通过对反射信号的接收、处理,以获得体内器官的图像。近年来,超声成像技术不断发展,出现了 3D 彩超、超声全息摄影、体腔内超声成像、彩色多普勒成像及超声生物显微镜等新的超声成像技术。
- PET图像:正电子发射断层扫描(PET)利用F18等放射性元素标记的示踪剂 衰变时发射的正电子信息成像,因此,PET图像是相应示踪剂放射性活度的度量, 能提供肿瘤生物学特性(如葡萄糖代谢、乏氧、增殖等)信息,其标准摄入值大小可用于临床辅助判别肿瘤良/恶性。PET能提供比CT、MRI更直观、更精确的可视化生物学与放射生物学特性信息。
- 病理图像:是指切取一定大小的病变组织,采用苏木精和曙红 (H & E) 等染色方法将切片组织做成病理玻片,然后用显微镜成像技术对微观的细胞和腺体成像。通过对病理图像进行分析,可探讨病变产生的原因、发病机理、病变的发生发展过程,从而做出病理诊断。
目前,临床医学图像分析深度学习研究对象主要可分为上述6类医学图像。
2.2 主要的医学图像分析任务
- 医学图像分类与识别
- 临床医生需要借助医学图像来辅助诊断人体是否有病灶,并对病灶的轻重程度进行量化分级,因此自动识别图像中的病灶区域和正常组织器官是医学图像分析的基本任务。
- 医学图像定位与检测
- 人体组织器官解剖结构和病灶区域的定位是临床治疗计划和干预流程中非常重要的预处理步骤,定位的精度直接影响治疗的效果。图像目标定位任务不仅需要识别图像中的特定目标,而且需要确定其具体的物理位置。图像目标检测任务则需要把图像中所有目标识别出来,且确定它们的物理位置和类别。
- 医学图像分割任务
- 图像分割是识别图像中感兴趣的目标区域内部体素以及外部轮廓,它是临床手术图像导航和图像引导肿瘤放疗的关键任务。
3、深度学习方法概述
- 机器学习算法通常分为有监督学习算法和无监督学习算法。在有监督学习算法中不仅把训练的数据输入给计算机,同时还把数据的标签也一并作为输入传给计算机,计算机进行学习之后,再给它传进新的未知的数据,它也能计算出该数据属于某种结果的概率,最后给你一个最接近正确的结果。由于计算机在学习的过程中不仅有训练数据,而且还有结果(标签),因此训练的效果往往都不错。与有监督学习不同的是,无监督学习的输入数据只有原始数据,没有标签,因此计算机无法准确地知道哪些数据属于哪些类,只能凭借强大的计算能力分析数据的特征,从而得到一定的成果,通常得到的是一些集合,集合内的数据在某些特征上相同,或相似。
3.1 神经网络
- 神经网络是一门重要的机器学习技术,在机器学习和认知领域。是一种模仿生物神经网络的结构和功能的数学模型或计算模型,用于对函数进行估计或近似。神经网络由大量的人工神经元联结进行计算。大多数情况下人工神经网络能在外界信息的基础上改变内部结构,是一种自适应系统,通俗的讲就是具备学习功能。现代神经网络是一种非线性统计性数据建模工具。典型的神经网络具有以下三个部分:
- 结构(Architecture):结构指定了网络中的变量和它们的拓扑关系。例如,神经网络中的变量可以是神经元连接的权重(weights)和神经元的激励值(activities of the neurons)。
- 激励函数(Activity Rule):大部分神经网络模型具有一个短时间尺度的动力学规则,来定义神经元如何根据其他神经元的活动来改变自己的激励值。一般激励函数依赖于网络中的权重(即该网络的参数)。
- 学习规则(Learning Rule):学习规则指定了网络中的权重如何随着时间推进而调整。这一般被看做是一种长时间尺度的动力学规则。一般情况下,学习规则依赖于神经元的激励值。它也可能依赖于监督者提供的目标值和当前权重的值。
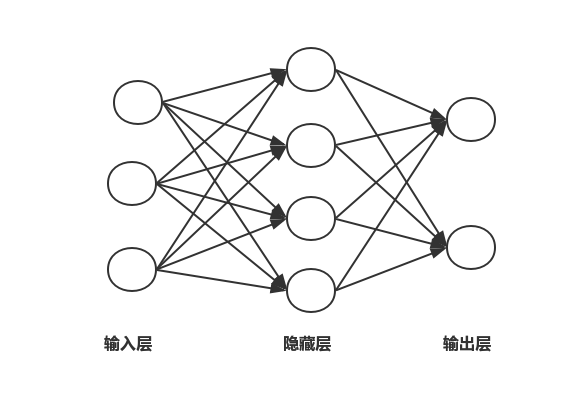
- 通常一个简单的神经网络包含3部分:输入层、隐含层和输出层。一般输入层和输出层的节点是固定的,隐含层可以自由指定,图1是一个简单的神经网络结构示意图,图中的圆圈代表神经元,箭头代表数据的流向,每根连线上面都对应一个不同的权重,权重是网络通过学习自己得到的。
- 每一个神经元都包含了输入、输出以及计算模型3个部分,可以把神经元看成一个计算与存储单元,计算是神经元对其的输入进行计算功能。存储是神经元会暂存计算结果,并传递到下一层。如图2所示是一个典型的神经元模型,包含3个输入,1个输出以及1个计算功能。
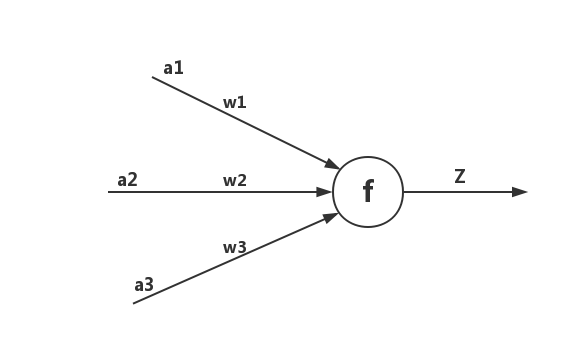
- 其中a表示输入,w表示权值,一个表示连接的有向箭头可以这样理解:在初端,传递的信号大小仍然是a,端中间有加权参数w,经过这个加权后的信号会变成aw,因此在连接的末端,信号的大小就变成了aw。在神经元处加权信号求和通过函数f就得到了输出Z,公式如下:
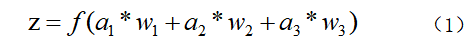
事实上,为了使神经元取得更好的效果,通常对每一个输入进行加权之后都会加上一个偏置b,此时Z的计算公式如下:
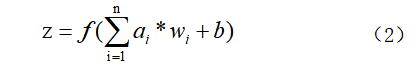
其中函数f又称为激活函数,常见的激活函数有sigmoid函数、tanh函数和ReLU函数等。
3.2 神经网络框架
1.深度神经网络(DNN)
- DNN是层数很深的全连接网络,层数决定了神经网络对数据的刻画能力:利用每层更少的神经元拟合更加复杂的函数。2006年Hinton利用预训练方法缓解了局部最优解问题,将隐含层推动到了7层,揭开了深度学习的浪潮。注意这里的“深度”并没有固定的定义,在语音识别领域,4层就认为是比较深的了;而在图像识别领域20层的模型屡见不鲜。2016年提出的highway network以及深度残差学习(deep residual learning)进一步杜绝了梯度消失达到了一百多层。
2.卷积神经网络(CNN)
- 卷积神经网络更改了神经网络的结构,不再是全连接的结构了,大量的减少了网络的参数;同时,通过参数共享进一步减少了网络参数。它考虑到了空间结构和局部特征,非常适用于图像处理领域,目前卷积神经网络在医学图像分析领域应用的最为广泛。例如对医学图像进行分割时,CNN可以简单的对图像中的每个像素进行分类,方法是在特定像素周围提取补丁。
3.循环神经网络(RNN)
- 鉴于深度学习在现实任务中的广泛适用性,它已经吸引了众多技术专家、投资者和非专业人员的关注。尽管深度学习最著名的成果是使用前馈卷积神经网络来解决计算机视觉问题,少数公众的注意力已经投入到使用递归神经网络来对时间关系进行建模。
在普通的全连接网络或者CNN中,每层神经元的信号只能向上传播,样本的处理在各个时刻独立,因此又称为前馈神经网络。而在RNN中,神经元的输出可以在下一个时间戳直接作用到自身;即第i层的神经元在m时刻的输入包含i-1层在该时刻的输出以及其自身在m-1时刻的输出。在此基础上发展出了长短期记忆LSTM网络。4.深度置信网络DBN
- 深度置信网络(deep belief networks,DBN)是一种包含多层隐单元的概率生成模型,可被视为多层简单学习模型组合而成的复合模型。可以作为深度神经网络的预训练部分,并为网络提供初始权重,再使用反向传播或者其他判定算法作为调优的手段。
- 虽然我们可以对深度学习的各种方法进行分类,但是广义上来说NN或者DNN是包含了其它各种变种方法的。在实际使用中往往也是多种结构的融合。
4、深度学习在医学图像中应的用
4.1 医学图像分类
- 医学图像分类可以分为图像筛查和目标或病灶分类。图像筛查是深度学习在医学图像分析领域中的最早应用之一, 是指将一个或多个检查图像作为输入, 通过训练好的模型对其预测, 输出一个表示是否患某种疾病或严重程度分级的诊断变量。图像筛查属于图像级分类,用来解决此任务的深度学习模型最初关注于 SAE、 DBN 及 DBM 网络和非监督预训练方法。研究主要集中在神经影像的分析上,如通过神经影像诊断是否患有老年痴呆症 (Alzheimer0s disease, AD) 或轻度认识功能障碍(Mild cognitive impairment, MCI)[90−92]。这些算法通常利用多模态图像作为输入,提取MRI、PET等模态中的互补特征信息。目前, CNN 正逐渐成为图像筛查分类中的标准技术,其应用非常广泛。如 Arevalo 等提出了乳腺癌诊断的特征学习框架, 采用CNN自动学习区分性特征,对乳房X线照片病变分类[3]。Kooi等比较了传统CAD中手动设计和自动CNN特征提取方法,两者都在约4.5万乳房X线图像的大数据集上训练,结果显示 CNN 在低灵敏度下优于传统CAD系统方法,且在高灵敏度下两者相当[4]。Spampinato等应用深度 CNN 自动评估骨骼骨龄[5]。另外,还有一些工作将CNN与RNN结合起来,如Gao等利用CNN 提取裂隙灯图像中的低层局部特征信息,结合RNN进一步提取高层特征,对核性白内障进行分级[6]。
- 目标或病灶的分类可以辅助医生对疾病进行诊断,如对乳腺病灶进行良恶性分类。其处理过程通常首先通过预处理方法识别或标记出的特定区域,然后再对特定区域进行目标或病灶分类。精确的分类不仅需要病灶外表的局部信息,而且还需结合其位置的全局上下文信息。CNN在目标或病灶的分类中也应用的非常广泛。Kawahara等采用多处理流CNN对皮肤病灶分类,其中每个流程处理不同分辨率的图像[7]。Jiao等利用CNN提取不同层次的深度特征,提高了乳腺癌的分类准确率[8]。Tajbakhsh 等就 CT 图像中检测肺结节且区分良性和恶性肺结节的任务,比较了大规模训练人工神经网络(Massive-training artificial neuralnetworks, MTANNs) 与 CNN 这两种端到端训练的人工神经网络的性能,其实验结果表明,只有使用较少训练数据时,MTANN的性能明显高于 CNN[9]。
4.2 医学图像定位与检测
- 准确地在医学图像中定位特定生物标记或解剖结构在临床治疗中具有非常重要的意义,直接关系到治疗效果的好坏。医学图像定位常需要分析3D体素信息。为了使用经典深度学习算法进行3D数据处理,一些方法将3D空间看成2D正交面的组合,这样可将定位任务转换成为分类任务,利用通用深度学习框架进行处理。如Yang等结合三个正交方向CNN的信息识别股骨末端的标记,标记3D位置定义为三个2D图块的交点[10]。Vos等通过将3D CT体积解析成2D形式,识别目标3D矩形包围盒,进而定位到感兴趣的心脏、主动脉弧和下降主动脉等解剖区域[11]。
- 而医学图像的感兴趣目标或病灶检测的关键是对每个像素进行分类。目前大多数基于深度学习目标检测系统采用CNN执行像素分类任务,之后采用某种形式的后处理方式得到目标。香港中文大学Chen等利用多个2D深层特征近似表达3D医学图像的特征,结合SVM分类器,实现敏感性加权图像(Susceptibility weighted imaging,SWI)自动检测脑微出血(Cerebral microbleeds, CMBs)[12]。还有少量工作采用其他深度学习方法来实现感兴趣目标或病灶检测, 如 Shin 等将SAE深度学习方法应用于MRI图像上检测腹部器官,先以非监督方式学习空间特征,然后基于兴趣点进行多器官检测[13]。
4.3 医学图像分割
- 医学图像分割的任务通常被定义为识别组成感兴趣对象的轮廓或内部的体素集,它是深度学习应用于医学图像分析领域的论文中最常见的主题。医学图像中器官及其子结构的分割可用于定量分析体积和形状有关的临床参数,如心脏的心室体积和收缩射出率。另一方面,在采用智能调强放疗技术对肿瘤进行治疗时, 危及器官勾画是制定放疗计划中非常重要的步骤之一,深度学习在此任务中应用非常广泛,主要应用于:组织病理学图像和显微镜图像分割;脑组织结构分割以及心脏心室分割等领域。
- 通过计算机分割来自手术和活检组织标本的图像特征可以帮助预测疾病侵袭性的程度,从而进行疾病诊断和分级。这些预测器的关键组成部分就是从组织病理图像挖掘的图像特征。目前绝大对数组织病理学图像和显微镜图像分割的方法都是基于CNN的。许多学者利用图块训练网络取得了非常优秀的分割结果。Ciresan等率先将深度CNN应用于医学图像分割,他们以滑窗方式在电子显微镜图像中分割出生物神经膜[14]。Kumar等利用基于块的CNN对H&E染色的病理学图像进行细胞核分割[15]。
- 脑组织结构的体积形态与很多的脑部神经疾病都息息相关。如抑郁症、阿耳茨海默氏病、精神分裂症和躁郁症等。因此通过计算机技术对脑组织结构的解剖结构进行研究在医学研究、临床诊断和治疗方面都起着十分重要的意义。例如Zhang 等将 T1、T2和FA (Fractional anisotropy) 三种模态图像作为输入,采用深度CNN网络解决婴儿GM、WM和CSF分割这一挑战性任务,从而对婴儿脑发育优劣程度作评估[16]。
- 从心脏MRI数据中分割出左心室是计算心室体积和收缩射出率等临床指标的重要步骤之一。Carneiro等采用基于DBN学习特征对左心室外观建模,利用监督学习模型在心脏超声波图像中自动分割出左心室[17],而Avendi等采用SAE 学习深度特征初步推断左心室的形状,再结合形变模型提高左心室分割的准确性和鲁棒性[18]。
- 从深度学习应用框架来看,目前大多数图像分割方法都是基于CNN的。许多学者利用基于图像块方式训练网络取得了很好的分割结果。
5、总结
- 综上所述,深度学习具有自动地从数据中学习深层次、更具鉴别性特征的能力,已应用于医学图像分析的多个研究领域,并取得了突破性进展。我们注意到,在大多数文献中,使用深度学习相关方法展示了其领先水平的性能,这已由医学图像分析的若干计算挑战赛结果证明;其次,云计算和多 GPU高性能并行计算技术的发展,使得深度学习从海量的医学图像大数据中学习深层特征成为可能; 最后,可公开访问的相关医学图像数据库的出现及多个医学图像分割挑战赛数据集,使得基于深度学习的分割算法能够得到有效验证。
- 我们相信,通过深度学习算法的不断改进,借助高性能并行计算技术的发展和日益改善的医学图像质量与不断增长的医学图像标记样本集,基于深度学习的医学图像分析将大有所为。
参考文献
[1]Zhang Lei, Zhang Yi. Big data analysis by infinite deep neural networks. Journal of Computer Research and Development, 2016, 53(1): 68−79.
[2]Litjens G, Kooi T, Bejnordi B E, Setio A A A, Ciompi F,Ghafoorian M, van der Laak J A W M, van Ginneken B,S´anchez C. A survey on deep learning in medical image analysis. Medical Image Analysis, 2017, 42(9): 60−88.
[3]Arevalo J, Gonz´alez F A, Ramos-Poll´an R, Oliveira J L,Lopez M A G. Representation learning for mammographymass lesion classification with convolutional neural networks. Computer Methods and Programs in Biomedicine,2016, 127: 248−257.
[4]Kooi T, Litjens G, van Ginneken B, Gubern-M´erida A,S´anchez C I, Mann R, den Heeten A, Karssemeijer N.Large scale deep learning for computeraided detection of mammographic lesions. Medical Image Analysis, 2017, 35:303−312.
[5]Spampinato C, Palazzo S, Giordano D, Aldinucci M,Leonardi R. Deep learning for automated skeletal bone age assessment in X-ray images. Medical Image Analysis, 2016,36: 41−51.
[6]Gao X T, Lin S, Wong T Y. Automatic feature learning to grade nuclear cataracts based on deep learning. IEEE Transactions on Biomedical Engineering, 2015, 62(11):2693-2701.
[7]Kawahara J, Hamarneh G. Multi-resolution-tract CNN with hybrid pretrained and skin-lesion trained layers. International Workshop on Machine Learning in Medical Imaging. Athens, Greece: Springer, 2016. 164−171.
[8]Jiao Z C, Gao X B, Wang Y, Li J. A deep feature based framework for breast masses classification. Neurocomputing, 2016, 197: 221−231.
[9]Tajbakhsh N, Suzuki K. Comparing two classes of end-toend machine-learning models in lung nodule detection and classification: MTANNs vs. CNNs. Pattern Recognition,2016, 63: 476−486.
[10]Yang D, Zhang S T, Yan Z N, Tan C W, Li K, Metaxas D.Automated anatomical landmark detection ondistal femur surface using convolutional neural network. In: Proceedings of the 12th International Symposium on Biomedical Imaging. New York, NY, USA: IEEE, 2015. 17−21.
[11]de Vos B D, Wolterink J M, de Jong P A, Viergever MA, Iˇsgum I. 2D image classification for 3D anatomy localization: employing deep convolutional neural networks.In: Proceedings of the 9784, Medical Imaging 2016: ImageProcessing. San Diego, California, US: SPIE, 2016, 9784:Article No. 97841Y.
[12]Chen H, Yu L Q, Dou Q, Shi L, Mok V C T, Heng PA. Automatic detection of cerebral microbleeds via deep learning based 3D feature representation. In: Proceedings of the 12th International Symposium on Biomedical Imaging (ISBI). New York, NY, USA: IEEE, 2015. 764−767.
[13]Shin H C, Orton M R, Collins D J, Doran S J, Leach M O. Stacked autoencoders for unsupervised feature learning and multiple organ detection in a pilot study using 4D patient data. IEEE Transactions on Pattern Analysisand Machine Intelligence, 2013, 35(8): 1930−1943.
[14]Cire¸san D C, Giusti A, Gambardella L M, Schmidhuber J. Deep neural networks segment neuronal membranes in electron microscopy images. In: Proceedings of the 25th International Conference on Neural Information Processing Systems. Lake Tahoe, Nevada: Curran Associates Inc., 2012. 2843−2851.
[15]Kumar N, Verma R, Sharma S, Bhargava S, Vahadane A,Sethi A. A dataset and a technique for generalized nuclear segmentation for computational pathology. IEEE Transactions on Medical Imaging, 2017, 36(7): 1550−1560.
[16]Zhang W L, Li R J, Deng H T, Wang L, Lin W L, Ji S W, Shen D G. Deep convolutional neural networks for multi-modality isointense infant brain image segmentation. NeuroImage, 2015, 108: 214−224.
[17]Carneiro G, Nascimento J C, Freitas A. The segmentation of the left ventricle of the heart from ultrasound data using deep learning architectures andderivative-based search methods. IEEE Transactions on Image Processing, 2012,21(3): 968−982.
[18]Avendi M R, Kheradvar A, Jafarkhani H. A combined deep-learning and deformable-model approach to fully automatic segmentation of the left ventricle in cardiac MRI.Medical Image Analysis, 2016, 30: 108−119.